Simon Boitard, Laurence Liaubet, Cyriel Paris, Katia Fève, Patrice Dehais, et al. Whole-genome sequencing of cryopreserved resources from French Large White pigs at two distinct sampling times reveals strong signatures of convergent and divergent selection between the dam and sire lines. Genetics Selection Evolution, 2023, 55, pp.13. 10.1186/s12711-023-00789-z hal-04016249
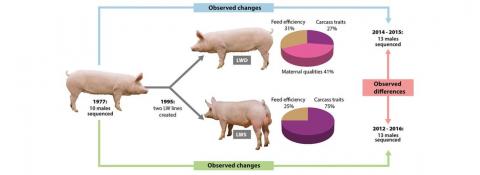
Les technologies récentes de génotypage et de séquençage des génomes permettent de décrire la diversité génétique actuelle d’un nombre croissant d’espèces et de races d’élevage, et de détecter l’évolution d’un grand nombre de régions génomiques sous l’effet de la sélection exercée depuis la domestication des espèces. Cependant, les processus de sélection sous-jacents sont très souvent méconnus : parmi les gènes situés dans une région détectée sous sélection, lequel a été sélectionné ? Quel caractère a-t-il permis de modifier ? Cette sélection est-elle liée à la domestication initiale de l’espèce, à la création de la race étudiée ou à son amélioration génétique récente ? A partir du cas de porcs Large White français, une équipe de scientifiques d’INRAE, de l’IFIP-Institut du porc et de Alliance R&D a montré que le séquençage de matériel biologique conservé en cryobanques permet de prendre en compte la diversité génétique ancestrale et de répondre ainsi plus finement à toutes ces questions.
L’étude détaillée de chaque région a également révélé que les signatures de sélection les plus marquées étaient souvent associées à des gènes dont la fonction biologique est particulièrement pertinente pour les caractères sélectionnés. Dans un de ces gènes, IGF2, on observe la sélection d’une mutation génétique affectant la croissance musculaire bien connue dans plusieurs races de porc dont le Large White.
